時系列データの扱い方 リサンプリングについて
毎時や毎日のように一定間隔でデータを集計する時系列データを扱う際、datetimeオブジェクトとして日時データを取り込むと日次から週次などの間隔を変更、再集計(リサンプリングといいます)が簡単にできます。
このブログでは政府が日々集計し公表している新型コロナワクチンの接種状況のデータを例にしてdatetimeオブジェクトの基本を解説しています。合わせてピボットテーブルでの集約方法も説明しています。双方を比較しながら、時系列データを使ったデータの分析、可視化の参考にしてください。
チートシート
| やりたいこと | 方法 |
|---|---|
改行区切りJson形式の接種データ(prefecture.ndjson)をデータフレームとして読み込む |
import jsonimport pandas as pdimport numpy as nprecords = map(json.loads, open('prefecture.ndjson'))df = pd.DataFrame.from_records(records) |
データ型が文字列のカラム名”date”をdatetime object にする |
df['date'].dtypes=> dtype('O')df['date']=pd.to_datetime(df['date'])df['date'].dtypes=> dtype('<M8[ns]') |
datetime object にしたcolumn名 'date' をインデックスにする |
df = df.set_index('date') |
| df1 を一回目接触(status =1)のデータフレーム df2を二回目接種(status=2)と2つに分割する |
df1 = df[df['status'] == 1]df2 = df[df['status'] == 2]print(df1.shape)print(df2.shape) |
| カラム名’date’の時系列データ ( summary_by_date)をdateオブジェクトとして読み込みデータフレーム名 df3とする |
df3 = pd.read_csv("summary_by_date.csv", parse_dates=["date"]) |
日次のデータ(df3)を週次にリサンプリングする(df3_w)。リサンプリング期間のデータは総和sum()する |
df3_w = df3.resample('W').sum() |
date 列をindexにして都道府県コードを列として一回目接種の接種回数countをsum計算するpivot table df4を作成する |
df4 = pd.pivot_table(df1, values='count', index=['date'], columns=['prefecture'], margins=False, aggfunc = np.sum) |
データは以下のリンクからダウンロードします。利用にあたっては、詳しい規約 に則って利用してください。
ダウンロードしたndjsonファイルは以下のカラムから構成されています。
'date', 'prefecture', 'gender', 'age', 'medical_worker', 'status', 'count'
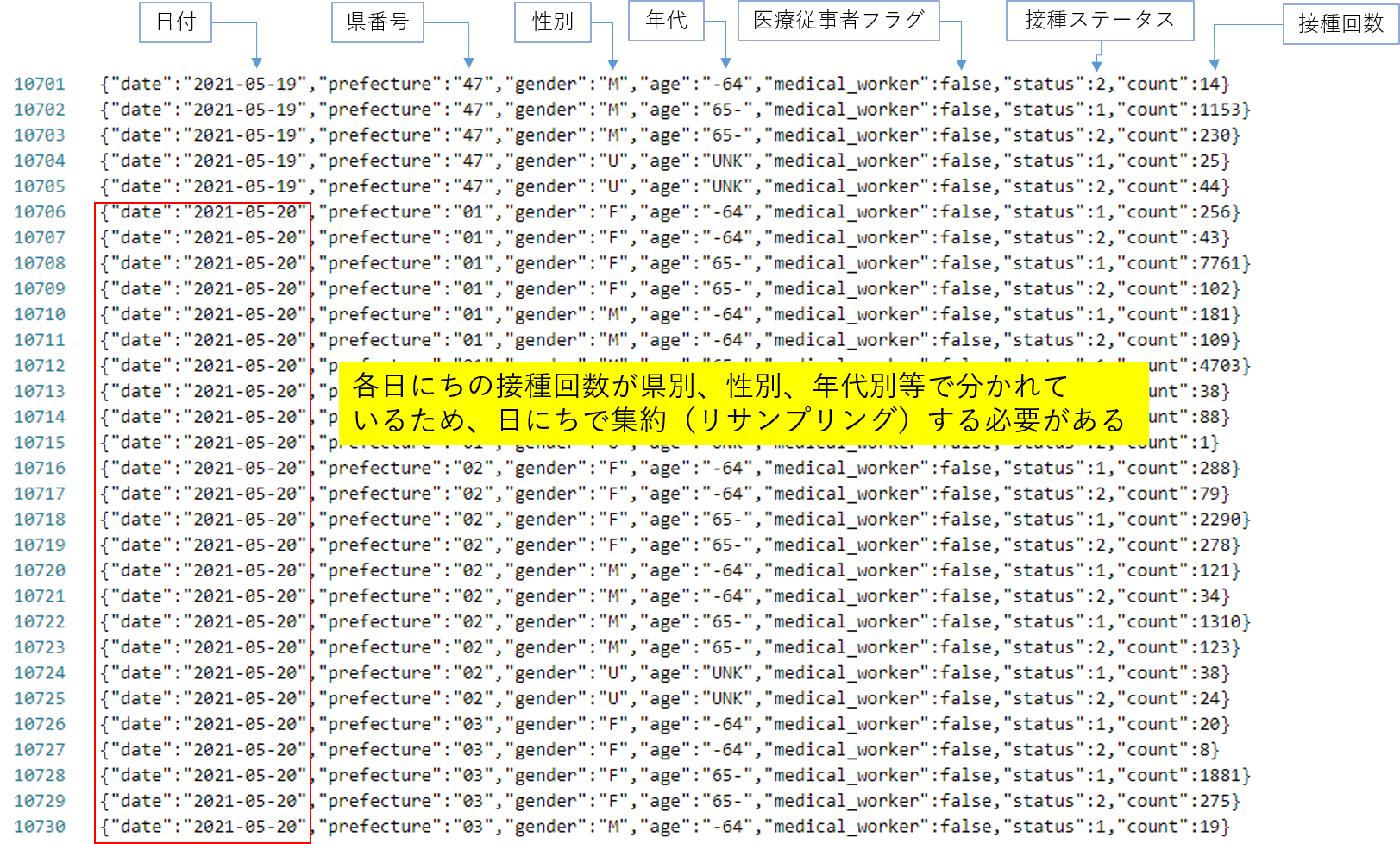
このデータの一部をテキストエディタで開けてみました。以下のとおり、各日にちの接種回数は都道府県別、性別、接種者群の年代を表す区分値等で細分化(一行に一系列化)されています。このデータからある日の接種回数を求めるにはその日を軸に集約する必要があります。
このポータルサイトには接種日別接種回数サマリーのデータも用意されていますので、無理に詳細データから集計する必要はありません。むしろ、答え合わせとして使うのも一案です。
集約する計算方法は、合計sum()と平均mean()が一般的です。もちろん、接種回数のような計算可能であることが前提です。例えば、性別であるとか年代のようなカテゴリカルデータの要素数の集計はできません。
リサンプリングの条件
時系列データはその間隔が毎時、日次、月次というように一定の間隔でデータを集計したものです。この間隔を変更することをリサンプリングといいます。
datetimeオブジェクトでリサンプリングを行うには
[Point!]
1.datetimeオブジェクトをインデックスとするデータフレームであること
リサンプリングの単位は週単位(’W’)、月単位(’M’)が一般できですが、二日おき(’2D’)、隔週(’2W’)も簡単に指定できます。時系列データの間隔を色々と変更して傾向分析も簡単にできます。
二日間の間隔にして、二日間の平均を計算したい場合は、以下になります。 Datetimeオブジェクトでインデックスとなっているデータフレームに対してリサンプリングをするので、カラム名とかの指定は不要です。二日おきは’2D’と指定します。
[Hint]
df_2d = df.resample(‘2D’).mean()
クロス集計表をpivot テーブル機能で作成
先のデータを今度は都道府県別に集計したいと思います。ピボットテーブルを使えば簡単に集約できます。日付をindexにして各都道府県別にそれぞれのカラム(感染者数、死者数)についてクロス集計表をpivot テーブル機能で作成します。
1
2
3
4
5
6
7
8
9
# pivot tableでやりたいこと:dateをindexにして各都道府県(prefecture)をカラムとするクロス集計表を作成する
# 集計要素は各都道府県の4/12から始まる各日の1回目接種(count)で集計し、都道府県コードのcolumnで展開する。
# pivot テーブルで作成したクロス集計表をさらにサンプリングするため、各行、各列の総数は出しません。 margins = Fales
#一日複数のエントリーがあるので、`aggfunc = np.sum`で総和をとします# df1 というデータフレームで受ける
df4 = pd.pivot_table(df9, values='count', index=['date'], columns=['prefecture'], margins=False, aggfunc = np.sum)
# NAN はゼロに変換します
f4 = df4.fillna(0)
# 整数にして小数点以下の表示省略させます。
df4 = df4.astype('int')
クロス集計表(データフレーム名 df4)は以下のような感じになるかと思います。
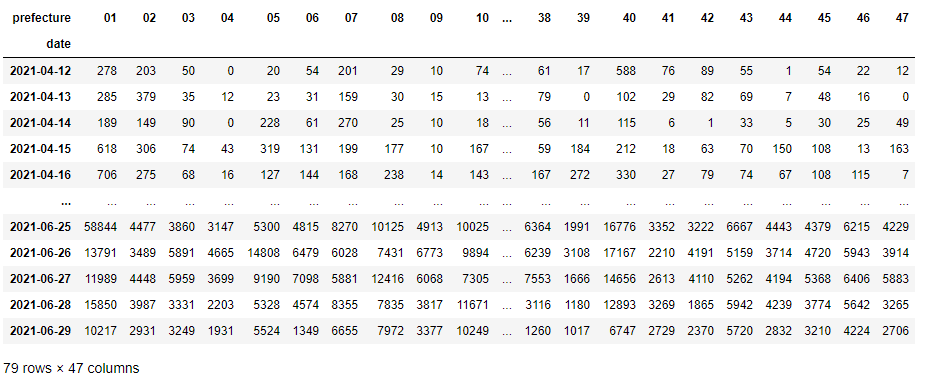
データは2021-06-29 までで79 rows × 47 columns すなわち、2021/4/12から2021/6/29の79日間について47都道府県別の一日あたりの集計表になります。
リサンプリング
試しに、日次を月次にリサンプリングして月次の接種回数のクロス集計表を作成します。
1
2
3
4
5
#時系列データの間隔を変更することをリサンプリングという
#元データ一日間隔を一か月間隔に変更するようにして、新しく一か月間隔(月初が起点)で集約されたデータについて総和をとる
df4_m = df4.resample('M').sum()
#データフレームの内容を表示します
df4_m
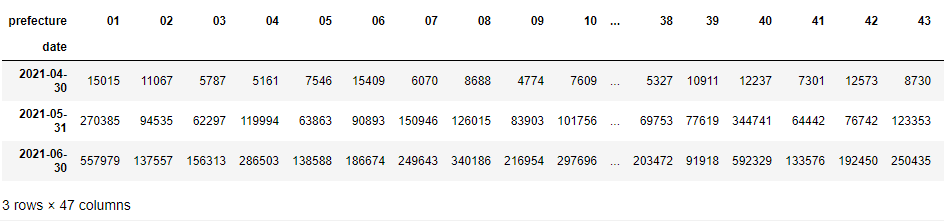
月次にリサンプリングした都道府県別のクロス集計表(データフレーム名 df4_m)は以下のような感じになるかと思います。
ひとこと
扱うデータの多くは時系列データの方も少なく無いと思います。今回ご紹介した新型コロナワクチンのオープンデータを練習台にして、datetimeオブジェクトのリサンプリングとピボットテーブルを使いこなし、時系列データ分析をマスターしてください。